
リンゴ果実特性の予測精度向上!ゲノム情報の統合で品種改良が加速!
リンゴの果実特性予測精度が向上!ゲノム情報の統合で品種改良が加速!
千葉大学国際高等研究基幹の南川舞准教授らの研究グループは、異なるゲノム解析システムから取得されたゲノム網羅的なDNAの違いのデータを統合する技術を開発しました。この技術により、過去に取得したデータも活用できるようになり、芽生えの段階におけるリンゴの多くの果実特性の予測精度が向上しました。さらに、糖度や食感のようないくつかの果実特性については、近親交配の程度の情報も考慮することで、予測のさらなる高精度化が可能であることを示しました。
この成果により、消費者や生産者の多様なニーズに的確に対応するリンゴの品種改良の高度化・効率化につながることが期待されます。本研究成果は、2024年7月8日に、学術誌Horticulture Researchでオンライン公開されました。
研究の背景
果樹は一般的に種子が発芽してから開花・結実するまでの期間が長く、交雑育種(品種改良)に長い年月を要します。また、穀物や野菜に比べて個体サイズが大きいために多数の個体を選抜対象にできず、新品種の育成が容易ではありません。しかし、近年注目されているゲノム情報を利用した育種法であるゲノミック選抜(Genomic Selection:GS)やゲノムワイド関連解析(Genome-Wide Association Study:GWAS)などの技術で、こうした果樹育種の障壁を取り除ける可能性があります。
近年、次世代シーケンサーの普及により、多数の品種間におけるゲノム網羅的なDNAの違い(一塩基多型:SNPs)を検出するためのゲノム解析システムは飛躍的に進歩しています。これまでGSやGWASでは、一つのシステムから得られたSNPs情報を使用することが主流でしたが、使用中のシステムが古くなった場合には、最新のシステムで解析し直す必要がありました。しかし、選抜時に廃棄された個体のDNAを再び得ることはできないため、過去のシステムで解析をした果樹育種の選抜用の集団を解析し直すことは困難でした。
研究グループは、異なるシステムにより得られたSNPs情報を上手く統合して活用する枠組みが必要だと考え、本研究では、世界的にポピュラーな果樹の一つであるリンゴを対象に、InfiniumとGRAS-Diシステムによりそれぞれ得られたSNPs情報の統合方法を検討し、大規模なデータの穴埋めを実施して統合したデータを用いた場合のゲノミック予測(Genomic Prediction:GP)やGWASの精度を評価しました。また、リンゴの交雑育種では少数の優れた起源品種や代表品種(例:ふじ)が、交雑用の親として繰り返し用いられてきており、近親交配が進んでいることが予想されます。近親交配の程度が大きくなると、樹勢や収量が低下することが知られています。このことから、近親交配の程度の情報も考慮した場合のGPやGWASの精度についても評価しました。
研究の成果
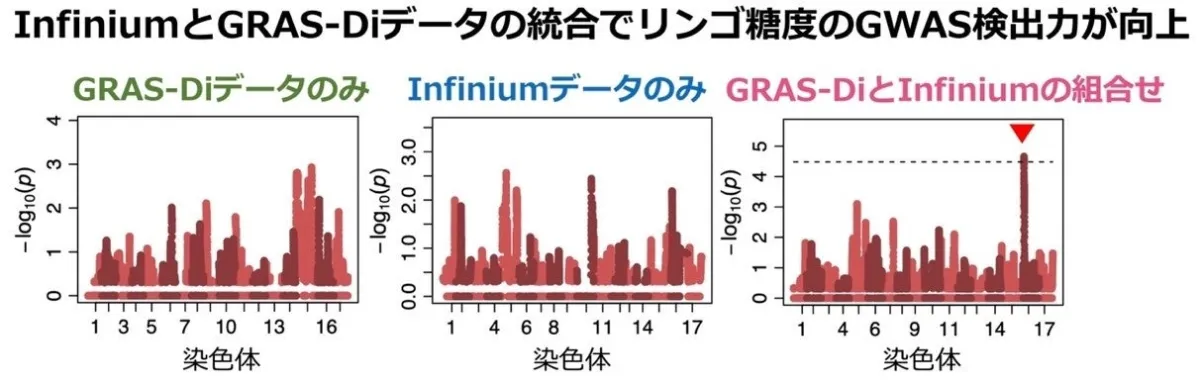
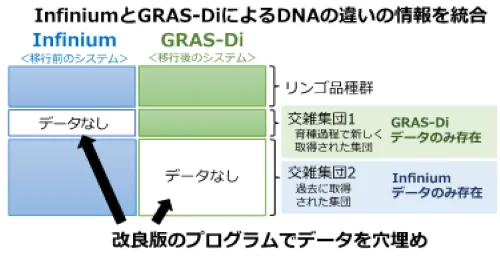
農研機構果樹茶業研究部門の盛岡研究拠点で栽培されているリンゴ品種群と交雑育種集団(計2,407個体)を解析に用いました。Infiniumは今後サービスが終了し使用が困難になることが予定されているため、将来GRAS-Diに移行することを仮定し、解析計画を立てました。Infiniumではリンゴ品種群と交雑集団2について、GRAS-Diではリンゴ品種群と交雑集団1について、DNAの違い(SNPs)のデータを取得しました。 GRAS-Diデータしかない交雑集団1の果実特性を予測するために、交雑集団2のInfiniumデータを利用できるよう、Beagleという既存のデータの穴埋めプログラムと自作のプログラムとの組合せによる改良版プログラムを用いて補完し、データの穴埋めを行いました。
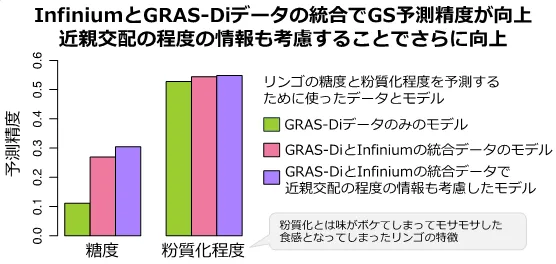
3種類の異なる情報と統計モデルのセットを用いて、交雑集団1におけるリンゴの糖度と、粉質化の程度に関するGP精度を比較した結果、GRAS-DiとInfiniumのそれぞれのシステムによって得られたSNPsデータを統合することで、GRAS-Diによって得られたSNPsのみを用いた場合よりもGP精度が向上することが多くの果実特性で明らかとなりました。いくつかの果実特性については、近親交配の程度の情報も考慮することで、GP精度がさらに向上しました。
次に、3種類の異なる情報のセットを用いて、GWASによる原因遺伝子領域の検出力を比較した結果、GRAS-DiとInfiniumのそれぞれのシステムによって得られたSNPsデータを統合することで、システム単体によって得られたSNPsのみを用いた場合よりも、GWASの検出力が向上することが多くの果実特性で明らかとなりました。GRAS-DiとInfiniumの統合データを使用した場合のみ、閾値である波線を超えるピークが検出されています。波線を超えた位置にあるSNPsは、糖度と統計的に有意な関連があることを示しています。
今後の展望
本研究により、異なるシステムから取得されたSNPsを組み合わせることで、将来ゲノム解析システムが変化していっても、過去のシステムで解析した個体のデータを活用できます。今回の調査で、過去のデータを統合することにより、芽生えの段階での、将来できるリンゴの果実特性の予測を高精度化できることが明らかになりました。また、GWASによる原因遺伝子領域の検出力も向上しました。開発した技術や方法論は、ナシやカンキツなどの他の果樹への応用も可能です。
今後は、3つ以上のシステムから取得された膨大なSNPs情報を統合する場合に備えて、その統合に用いるプログラムのさらなる高速化・高精度化を目指していく予定です。

トピックス(科学)










【記事の利用について】
タイトルと記事文章は、記事のあるページにリンクを張っていただければ、無料で利用できます。
※画像は、利用できませんのでご注意ください。
【リンクついて】
リンクフリーです。